|
 |
- Search
| J. Conserv. Sci > Volume 34(1); 2018 > Article |
|
초 록
Î≥∏ Ïó∞͵¨Îäî Î∂ÄÏÇ∞ Î∂Å͵¨ ÌôîΙÖÎèô Ï°∞ÏÑÝÏãúÎåÄ Î∂ÑΨò͵∞ÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκàΕº ÎåÄÏÉÅÏúºÎ°ú Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Î∂ÑÏÑùÏùÑ ÏàòÌñâÌïú Í≤∞Í≥ºÏù¥ Îã§. Ïã§Î¶¨Ïπ¥ Ï∂îÏ∂úÎ≤ïÏùÑ ÏǨÏö©ÌïòÏó¨ ÌÜÝÍ¥ëΨòÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκà 8Í∞úÏ≤¥Ïùò DNAΕº Î∂Ñζ¨ÌïòÏòÄÍ≥Ý, ÎØ∏ÌÜÝÏΩòÎìúζ¨ÏïÑ DNA Í≥ºÎ≥ÄÏù¥Î∂ÄÏúÑ Î∂ÑÏÑùÏùÑ ÌܵÌï¥ Î™®Í≥Ñ ÏúÝÏó∞Í¥ÄÍ≥Ñ Ïó¨Î∂ÄΕº ÌôïÏù∏ÌïòÏòÄÎã§. Î∂ÑÏÑù Í≤∞Í≥º ÌÜÝÍ¥ëΨò 11Ìò∏, 21Ìò∏, 26Ìò∏ÏóêÏÑú Ï∂úÌÜÝÎêú ÌîºÏû•Ïûê 3ΙÖÏùò ÌïòÌîåΰúÌÉÄÏûÖ Ïù¥ ÎèôÏÝïÎêòÏóàÏúºÎ©∞ HaploGrep 2 ÌîÑΰúÍ∑∏Îû®ÏóêÏÑú A5a, D4aÏôÄ M4"67+16311 ÌïòÌîåΰúÍ∑∏ΣπÏúºÎ°ú Î∂ÑΕòÎêòÏóàÎã§. ÌïòÌîåΰúÍ∑∏ΣπÏù¥ ÎèôÏÝïÎêú 3Í∞úÏ≤¥Îäî Í∞ôÏùÄ Î≥ÄÏù¥ÌòïÏùÑ Í≥µÏúÝÌïòÏßÄ ÏïäÏúºÎØÄΰú ÌîºÏû•Ïûê Í∞Ñ Î™®Í≥Ñ ÏπúÏó∞Í¥ÄÍ≥ÑÎäî ÏóÜÎäî Í≤ÉÏúºÎ°ú ÌôïÏù∏ÎêòÏóàÎ㧠. Ïù¥Î≤à Ïó∞͵¨Îäî ÏòÅÎÇ®ÏßÄÏó≠ÏóêÏÑú Ï∂úÌÜÝÎêú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÏùò Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Î∂ÑÏÑùÏùò Ï≤´ ÏǨΰÄΰúÏÑú Í≥ºÍ±∞ ÌïúÎ∞òÎèÑÏóê ÏÇ¥ÏïòÎçò ÏòõÏǨÎûåÎì§Í≥º ÌòÑÎåÄÏù∏Îì§ Ïùò ÏúÝÏÝÑÌïôÏÝŠͥÄÍ≥ÑΕº Ïù¥Ìï¥ÌïòÍ∏∞ ÏúÑÌïú Í∏∞Ï¥à ÏûêΣåΰú ÌôúÏö©ÌïÝ Ïàò ÏûàÏùÑ Í≤ÉÏúºÎ°ú Í∏∞ÎåÄÎêúÎ㧠.
ABSTRACT
The analysis of ancient DNA extracted from archaeological bones has become an important research tool in palaeogenetics and anthropology. Eight human skeletal remains of the Joseon dynasty, excavated from Hwamyeong-dong, were used in this study. DNA was extracted from bone powder using a silica-based protocol. The isolated DNA was analyzed by the sequencing variation of hyper-variable region of the mitochondrial DNA. In the present study, 3 human remains were identified into mtDNA haplogroups including the A 5a, D4a, and M4"67+16311 groups, using HaploGrep 2 program. The identified haplotypes of the 3 samples have been confirmed that the specimens in the tombs were not related by the maternal line. This is the first analysis of human skeletal remains of the Joseon dynasty excavated in Busan. Date from the analysis of human remains from the Joseon dynasty are considered as the basis for understanding the genetic relationship between modern and ancient humans of the Korean peninsula.
ϵúÍ∑º ÏúÝÏÝÅÏßÄÏóêÏÑú Ï∂úÌÜÝÎêòÎäî ÏòõÏǨÎûå κàÏóê ÎåÄÌïú Í¥ÄÏã¨Ïù¥ ϶ù Í∞ÄÌï®Ïóê Îî∞Îùº κàÏóê ÎÇ®ÏïÑÏûàÎäî DNAÎÇò Îã®Î∞±Ïßà Ïó∞͵¨Î•º ÏúÑÌï¥ ÏÉù ΨºÌïô, ÌôîÌïôÏÝÅ Î∂ÑÏÑù Í∏∞Î≤ïÏù¥ ÌôúÏö©ÎêòÍ≥Ý ÏûàÎã§. ÏǨÎûåκàÎäî ÌîºÏû•Ïûê Ïùò ÏúÝÏÝÑÏÝÅ ÌäπÏßïÏù¥ÎÇò ÏѱÎ≥ÑÎøêÎßå ÏïÑÎãàÎùº ÌîºÏû•Ïûê Í∞ÑÏùò ÌòàÏó∞관 Í≥ÑΕº ÌååÏïÖÌïÝ Ïàò ÏûàÏúºÎ©∞ Îçî ÎÇòÏïÑÍ∞Ä ÏßàÎ≥ë ÏúÝΨ¥ÎÇò ÏòÅÏñë ÏÉÅÌÉúΕº ÌååÏïÖÌïòÍ∏∞ ÏúÑÌïú Ïó∞͵¨ÏóêÎèÑ ÌôúÏö©ÎêÝ Ïàò ÏûàÎã§(Willerslev and Cooper, 2005). ͵≠ÎÇ¥ÏóêÏÑúÎäî ÎÇòÏ£º Î≥µÏïîζ¨ ÏúÝÏÝÅÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκàÏùò DNA Î∂ÑÏÑù(Lee et al., 1999)ÏùÑ ÏãúÏûëÏúºÎ°ú Í≤ΩÏÇ∞ ÏûÑ ÎãπÎèô Í≥ÝÎ∂Ñ͵∞Í≥º Ï°∞ÏòÅÎèô Í≥ÝÎ∂Ñ͵∞ÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκàΕº ÎåÄÏÉÅÏúº ΰú Ï≤¥ÏßàÏù∏ΕòÌïôÏÝÅ Î∂ÑÏÑùÍ≥º Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Î∂ÑÏÑù Î∞è ÏïàÏÝïÎèôÏúÑÏõê ÏÜå Î∂ÑÏÑù Í≤∞Í≥ºÍ∞Ä Î≥¥Í≥ÝÎêú Î∞î ÏûàÎã§(Lee et al., 2008a; Shin and Lee, 2009; Ha, 2011). Ïù¥Ïô∏Ïóê ÍπÄÌè¨ Ïû•Í∏∞ÎèôÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨ ÎûåκàÏùò ÏúÝÏÝÑÌïôÏÝÅ Î∂ÑÏÑùÍ≥º κàÏóê ÎÇ®ÏïÑÏûàÎäî ÎØ∏ÏÉùΨºÏùò Î∂ÑÌè¨Í∞Ä ÎºàÏùò Î≥¥Ï°¥ ÏÉÅÌÉúÏóê ÎØ∏ÏπòÎäî ÏòÅÌñ•Ïóê ÎåÄÌï¥ Í≥ÝÏ∞∞ÌïòÏòÄÎã§(Cho et al., 2010; Seo et al., 2014). ÎòêÌïú Ìò∏ÎÇ®ÏßÄÏó≠ÏóêÏÑú Ï∂úÌÜÝÎêú Ï°∞ ÏÑÝÏãúÎåÄ ÏǨÎûåκàÏùò ÏïàÏÝïÎèôÏúÑÏõêÏÜå Î∂ÑÏÑùÍ≥º Ïä§Ìä∏ΰÝÌä¨ ÎèôÏúÑÏõêÏÜå Î∂ÑÏÑù Îì± Îã§ÏñëÌïú ÎèôÏúÑÏõêÏÜå Î∂ÑÏÑùÍ∏∞Î≤ïÏùÑ ÌôúÏö©Ìïú Ïó∞͵¨Îì§Ïù¥ ÏßÑ ÌñâÎêòÍ≥Ý ÏûàÎã§(Park et al., 2015; Shin et al., 2015).
ÏòõÏǨÎûå κàÏùò ÏúÝÏÝÑÌïôÏÝÅ Ïó∞͵¨Îäî ÏúÝÏÝÅÏßÄÏùò ÏѱÍ≤© Í∑úΙÖÏùÑ ÏúÑ Ìï¥ ÌîºÏû•ÏûêÏùò Í∞úÏù∏ ÏãùÎ≥ÑÍ≥º Í∞ôÏù¥ ÎØ∏ÏãúÏÝŠΙ©ÏÝÅÏúºÎ°ú ÌñâÌï¥ÏßÄÍ∏∞ ÎèÑ ÌïúÎã§. ÌïòÏßÄÎßå Í∂ÅÍ∑πÏÝÅÏúºÎ°ú ÏòõÏǨÎûå κàÏùò ÏúÝÏÝÑÏÝïÎ≥¥Îäî Ïù∏Εò Ïùò ÏßëÎã®ÏúÝÏÝÑÌïôÏÝÅ Î≥ÄÌôîΕº ÌܵÏãúÏÝÅÏúºÎ°ú ÎπÑ͵ê Î∂ÑÏÑùÌïÝ Ïàò ÏûàÎäî Ïã§Ï¶ùÏÝÅ ÏûêΣåÎùºÎäî ÏÝêÏóêÏÑú ÌïôÏàÝÏÝÅ ÏùòÏùòÍ∞Ä ÌŨÎã§. ÌäπÌûà Ï°∞ÏÑÝÏãú ÎåÄ ÏǨÎûåκàÎäî Ïù¥ÏÝÑ ÏãúÎåÄÏóê ÎπÑÌï¥ Ï∂úÌÜÝ Í∞úÏ≤¥ ÏàòÍ∞Ä ÎßéÍ≥Ý Î≥¥Ï°¥ ÏÉÅÌÉúÍ∞Ä ÏñëÌò∏Ìïú Í≤ΩÏö∞Í∞Ä ÎßéÏúºÎØÄΰú ÎãπÏãúÏùò Ïù∏ΕòÏúÝÏÝÑÌïôÏÝÅ ÌäπÏѱ ÏùÑ Ïó∞͵¨ÌïòÍ∏∞ ÏúÑÌïú ÎåÄÏÉÅÏúºÎ°ú ÎߧÏö∞ ÏúÝÏö©ÌïòΩ∞, ÌòÑÎåÄ ÌïúÎ∞òÎèÑ Ïù∏ Εò ÏßëÎã®Ïùò ÌòïÏѱÍ≥º ÏßëÎã®ÏúÝÏÝÑÌïôÏÝÅ Î≥ÄÌôî ÏñëÏÉÅÏùÑ Íµ¨Ï≤¥ÏÝÅÏúºÎ°ú ÌååÏïÖÌïòÍ∏∞ ÏúÑÌïú Í∞Ä͵ê Ïó∞͵¨Î°úÏÑú ϧëÏöîÌïú Í∞ÄÏπòÍ∞Ä ÏûàÎã§.
ÌòÑÏû¨ÍπåÏßÄ ÍπÄÌè¨ Ïû•Í∏∞Îèô, ÍπÄÌè¨ ÌïôÏö¥Î¶¨, ÏÑúÏ≤ú Ïò•ÎǮζ¨, ÏïÑÏÇ∞ 명 Ïïîζ¨ Îì± Í≤ΩÍ∏∞¬∑Ï∂©Ï≤≠ ÏßÄÏó≠ÏóêÏÑú Ï∂úÌÜÝÎêú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÏùò Î∂ÑÏûê ÏúÝÏÝÑÌïôÏÝÅ Ïó∞͵¨ Í≤∞Í≥ºÎäî Îã§Ïàò ÌôïÏù∏ÎêòÏóàÏßÄÎßå, ÏòÅÎÇ® ÏßÄÏó≠ÏóêÏÑú Ï∂ú ÌÜÝÎêú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÏùò Î∂ÑÏÑù Í≤∞Í≥ºÎäî ÏïÑÏßÅÍπåÏßÄ Î≥¥Í≥ÝÎêú Î∞îÍ∞Ä ÏóÜÎã§(Jee et al., 2008; Kang et al., 2010; Seo et al., 2014; Jin et al., 2015; Kim et al., 2015). Î≥∏ Ïó∞͵¨Îäî Î∂ÄÏÇ∞ Î∂Å͵¨ ÌôîΙÖÎèô Ï°∞ ÏÑÝÏãúÎåÄ Î∂ÑΨò͵∞ÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκà 8ÏÝêÏóê ÎåÄÌïú ÎØ∏ÌÜÝÏΩòÎìúζ¨ÏïÑ DNA Î∂ÑÏÑù(mitochondrial DNA; mtDNA)ÏùÑ ÌܵÌïòÏó¨ Î≥ÄÏù¥ÌòïÏùÑ Í≤∞ÏÝïÌïòÍ≥Ý ÌïòÌîåΰúÍ∑∏ΣπÏùÑ ÎèôÏÝïÌïòÍ∏∞ ÏúÑÌïú Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Ïã§ÌóòÏù¥ ÏàòÌñâÎêòÏóàÎã§. ÎòêÌïú Î∂ÑÏÑù Í≤∞Í≥ºÎ•º ÌÜÝÎåÄΰú Í∑ºÏÝëÌïú ÏúÑÏπòÏóê ÎߧÏû•Îêú ÌîºÏû•Ïûê Í∞ÑÏùò Ι®Í≥Ñ ÏúÝÏó∞Í¥ÄÍ≥Ñ Ïó¨Î∂ÄΕº Í≤ÄÌÜÝÌïòÏòÄÎäîÎç∞ Ïù¥Îäî ÏòÅÎÇ® ÏßÄÏó≠ÏóêÏÑú Ï∂úÌÜÝÎêú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκà Ïó∞͵¨Ïùò Ï≤´ ÏǨΰÄÏù¥Îã§.
Ï°∞ÏǨ ÎåÄÏÉÅ ÏßÄÏó≠ÏùÄ Î∂ÄÏÇ∞ ÌôîΙÖÎèô Ïô∏Í≥Ω ÎèÑΰúÍ∞úÏѧÎ∂ÄÏßÄÏùò Ïùº ÌôòÏúºÎ°ú 1914ÎÖÑÏóê Î∂ÄÏÇ∞Í¥ëÏó≠Ïãú ÎèôÎûò͵∞ÏúºÎ°ú Ìé∏ÏûÖÎêòÍ∏∞ ÏÝÑÍπåÏßÄ Îäî Í≤ΩÏÉÅÎÇ®ÎèÑ ÏñëÏÇ∞Ïãú ÏùºÏõêÏóê ÏÜçÌï¥ ÏûàÏóàÎã§. ÏñëÏÇ∞ ÌïòÎ∂ÅÏÝï Í≥ÝÎ∂Ñ Íµ∞ÏóêÏÑú Ï∂úÌÜÝÎêú ÌÉÄÏÝúÏÑùÍ∏∞ ÏúÝΨºÎ°ú Î≥¥ÏïÑ Íµ¨ÏÑùÍ∏∞ÏãúÎåÄÎ∂ÄÌÑ∞ ÏǨÎûå Îì§Ïù¥ ÏÇ¥ÏïòÏùÑ Í≤ÉÏúºÎ°ú Ï∂îÏÝïÎêòΩ∞ ÏãÝÏÑùÍ∏∞ÏãúÎåÄ Ï£ºÍ±∞ÏßÄÏôÄ Ìå®Ï¥ùÏù¥ Î∂ÄÏÇ∞Ïãú Î∂Å͵¨ Í∏àÍ≥°ÎèôÏóê ÏúÑÏπòÌïú Ïú®Î¶¨Ìå®Ï¥ùÏóêÏÑú ÌôïÏù∏ÎêòÏóàÎã§. Ïù¥Ï≤òÎüº Ï°∞ÏǨÏßÄÏó≠Ïùò Ï£ºÎ≥ÄÏóêÎäî ÏãÝÏÑùÍ∏∞ÏãúÎåÄÎ∂ÄÌÑ∞ Ï°∞ÏÑÝÏãúÎåÄÏóê Ïù¥Î•¥Í∏∞ÍπåÏßÄ Îã§ÏñëÌïú Ïó∞ÎåÄÏùò ÏúÝÏÝÅÏù¥ Î∂ÑÌè¨ÌïúÎã§.
Î∂ÄÏÇ∞ ÌôîΙÖÎèô ÏúÝÏÝÅÏùÄ 2010ÎÖÑ 1ÏõîÎ∂ÄÌÑ∞ 6ÏõîÍπåÏßÄ (Ïû¨)Ìïú͵≠ Ψ∏ΨºÏó∞͵¨ÏõêÏóêÏÑú Î∞ú͵¥Ìïú Ï°∞ÏÑÝÏãúÎåÄ Î∂ÑΨò͵∞ÏúºÎ°ú ÏöîÏßĬ∑ÎÇôÎèôÍ∞ï ÏñëÏïàÏóê Î∂ÑÌè¨ÌïòÎäî ÎÇòΣ®ÌÑ∞ÏôÄ Í∞ÄÏïºÏßÑÏǨ Îì±Ïù¥ ÏúÑÏπòÌï¥ ÏûàÎã§. Ï°∞ ÏǨ Í≤∞Í≥º Ï°∞ÏÑÝÏãúÎåÄ ÌÜÝÍ¥ëΨò 26Í∏∞Í∞Ä ÌôïÏù∏ÎêòÏóàÎäîÎç∞ ÏÝïÎ∞ÄÎ∞ú͵¥ Ï°∞ÏǨÍ≥ºÏÝï ϧë 1͵¨Ïó≠ 2Ìò∏, 2͵¨Ïó≠ 11Ìò∏, 14Ìò∏, 16Ìò∏, 17Ìò∏, 21 Ìò∏, 23Ìò∏ ÌÜÝÍ¥ëΨòÏôÄ 2Ï∞® Ïãú͵¥Ï°∞ÏǨΕº ÌܵÌï¥ 3͵¨Ïó≠ 24Ìò∏, 26Ìò∏ ÌÜÝÍ¥ëΨò Îì± 9Í∏∞ÏóêÏÑú ÏǨÎûåκàÍ∞Ä Ï∂úÌÜÝÎêòÏóàÎã§. Î∂ÑΨò Î≥Ñ ÏǨÎûåκà Ï∂úÌÜÝ ÌòÑÌô©ÏùÄ Îã§ÏùåÍ≥º Í∞ôÎã§. 2Ìò∏ ΨòÏóêÏÑú ÏïÑÎûòÌѱκàÏóê ÏπòÏïÑ(Ïïû Îãà, ÏûëÏùÄÏñ¥Í∏àÎãà, ÏÜ°Í≥≥Îãà, ÌÅ∞Ïñ¥Í∏àÎãà Îì±) 7Í∞úÏôÄ Î®∏ζ¨Îºà ÏùºÎ∂ÄÍ∞Ä Ï∂úÌÜÝÎêòÏóàÏúºÎÇò Î∂ÄÏãùÏúºÎ°ú Ïù∏Ìï¥ ÏûîÏ°¥ÏÉÅÌÉúÍ∞Ä Î∂àÎüâÌïòÏòÄÍ≥Ý 11Ìò∏ ΨòÏóêÏÑúÎäî ή∏ζ¨Îºà Ï°∞Í∞ÅÍ≥º Ï¢åÏö∞Ï∏° Îã§Î¶¨ÎºàÍ∞Ä ÌôïÏù∏ÎêòÏóàÎã§. 14 Ìò∏ ΨòÎäî ή∏ζ¨Îºà Î∞è Ïö∞Ï∏° ÌåîκàÏôÄ Ï¢åÏö∞Ï∏° Îã§Î¶¨Îºà Îì±Ïù¥ ÏùºÎ∂Ä ÏûîÏ°¥ÌïòÍ≥Ý ÏûàÏóàÏúºÎ©∞ 16Ìò∏ ΨòÎäî ή∏ζ¨Îºà Î∞è Ïö∞Ï∏° ÌåîκàÏôÄ Îã§Î¶¨ κà, Ïö∞Ï∏° Í∞àÎπÑκà, Ï¢åÏö∞Ï∏° ÎÑôÎã§Î¶¨Îºà, Ïö∞Ï∏° Îã§Î¶¨Îºà ÏùºÎ∂ÄÍ∞Ä ÎÇ® ÏïÑÏûàÏóàÏúºÎÇò ÏûîÏ°¥ÏÉÅÌÉúÎäî ÏñëÌò∏ÌïòÏßÄ Î™ªÌïòÏòÄÎã§. 17Ìò∏ ΨòÏóêÏÑú Îäî ή∏ζ¨ÎºàÏôÄ Îã§Î¶¨ÎºàÍ∞Ä ÏùºÎ∂Ä ÎÖ∏Ï∂úÎêòÏóàÍ≥Ý 21Ìò∏ ΨòÏóêÏÑúÎäî ή∏ ζ¨Îºà, ÎπóÏû•Îºà, Í∞àÎπÑκà, ÏúÑÌåîκà, Ï≤ôÏ∂îκà, ÎÑôÎã§Î¶¨Îºà, ÏÝïÍ∞ïκà, ÏÜêÍ∞ÄÎùΩκà Îì±Ïù¥ ÏûîÏ°¥ÌïòÍ≥Ý ÏûàÎäî ÏÉÅÌÉúΰú 2͵¨Ïó≠ Ï°∞ÏǨÏßÄÏó≠ÏóêÏÑú Í∞ÄÏû• Ïò®ÏÝÑÌïú ÏÉÅÌÉúÏùò ÏǨÎûåκàÍ∞Ä Ï∂úÌÜÝÎêòÏóàÏúºÎ©∞ 23Ìò∏ ΨòÎäî ή∏ ζ¨Îºà ÏùºÎ∂ÄÎßå ÌôïÏù∏ÎêòÏóàÎã§. 3͵¨Ïó≠ 24Ìò∏ ΨòÏóêÏÑúÎäî Ï¢åÏö∞ Îã§Î¶¨ κàÏôÄ Î®∏ζ¨Îºà ÏùºÎ∂ÄÍ∞Ä ÎÖ∏Ï∂úÎêòÏóàÍ≥Ý 26Ìò∏ ΨòÎäî ή∏ζ¨Îºà, Í∞àÎπÑκà, Ï≤ôÏ∂îκà, ÏúÑÌåîκà, ÎÖ∏κà, Ïûêκà, Î≥ºÍ∏∞κà, Ï¢åÏö∞ ÎÑôÎã§Î¶¨Îºà, ÏÝïÍ∞ï κà, Ï¢ÖÏïÑζ¨Îºà, ÏÜêÍ∞ÄÎùΩκà Îì±Ïù¥ Ïò®ÏÝÑÌûà ÎÇ®ÏïÑÏûàÎäî ÏÉÅÌÉúΰú ÌôïÏù∏ ÎêòÏóàÎã§(The Korea Archaeology and Art History Research Institute, 2012).
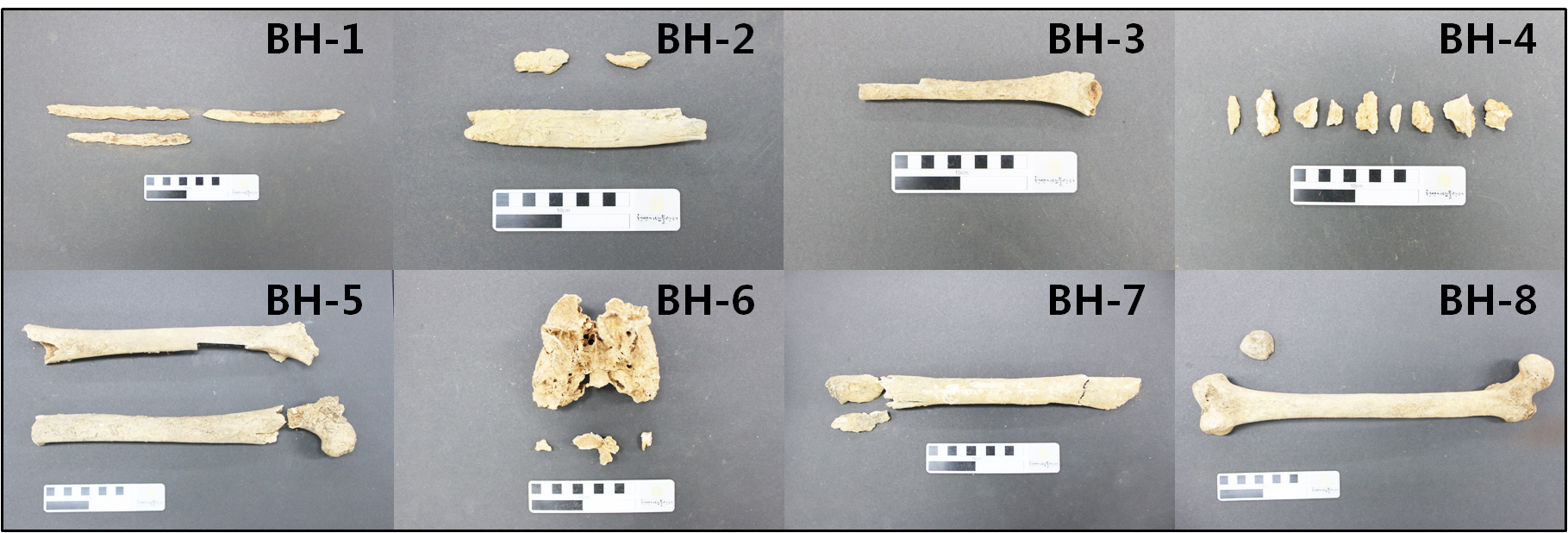
Î∂ÑΨò͵∞Ïùò Ï°∞Ïѱ ÏãúÍ∏∞Îäî Ψ¥Îç§ÏóêÏÑú Ï∂úÌÜÝÎêú Î∂ÑÏ≤≠ÏûêÏôÄ Î∞±Ïûê Îì±ÏùÑ ÌܵÌï¥ 15ÏÑ∏Í∏∞ ÌõÑÎ∞òÎ∂ÄÌÑ∞ 16ÏÑ∏Í∏∞ ϧëÎ∞òÏúºÎ°ú Ï∂îÏÝïÌïòÍ≥Ý Ïûà ÏúºÎ©∞, Ïù¥Îäî ÎãπÏãú Í≤ΩÏÉÅÎèÑ ÏßÄÏó≠ÏóêÏÑú Ï∂ïÏ°∞Îêú ÌÜÝÍ¥ëΨòÏùò ͵¨Ï°∞ Î∞è Î∂ÄÏû• ÏñëÏÉÅÍ≥º ÏúÝÏǨÌïú Í≤ÉÏúºÎ°ú ÏǨΣåÎêúÎã§. 16Ìò∏ ÌÜÝÍ¥ëΨò ÌîºÏû•Ïûê Îäî ή∏ζ¨ÎºàÏôÄ ÏôºÏ™Ω ÎπóÏû•Îºà ÏǨÏù¥ÏóêÏÑú Ìù¨ÎÖïÏõêÎ≥¥(ÁÜôÂØßÂÖÉÂØ∂) 1 ÏÝêÏù¥ Ï∂úÌÜÝÎêòÏóàÎäîÎç∞, Ï∂úÌÜÝ ÏúÑÏπòΕº Í≥ÝÎݧÌñàÏùÑ Îïå Ïû•Î°Ä Ïãú ÏóºÏäµ (Ô¶•Ë•≤) Í≥ºÏÝïÏóêÏÑú Î∞òÌï®(È£ØÂê´)ÏùÑ ÏßÑÌñâÌïú Í≤ÉÏúºÎ°ú Ï∂îÏÝïÎêúÎã§. Îòê Ìïú 14Ìò∏, 21Ìò∏, 26Ìò∏ ÌîºÏû•ÏûêÏùò Í≤ΩÏö∞ Í≥®Î∞ò ÏúÑÏπòÏóêÏÑú ÏÜêÍ∞ÄÎùΩ ÎºàÍ∞Ä ÌôïÏù∏ÎêòÏóàÏúºÎØÄΰú ÏóºÏäµ ÌõÑ ÎߧÏû•Îêú Í≤ÉÏúºÎ°ú ÌåêÎã®ÎêúÎã§(The Korea Archaeology and Art History Research Institute, 2012). Î∂ÑÏûêÏúÝÏÝÑÌïô Ïó∞͵¨Îäî 2Ìò∏ ÌÜÝÍ¥ëΨòÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκàΕº ÏÝúÏô∏ Ìïú 8Í∞úÏ≤¥Î•º ÎåÄÏÉÅÏúºÎ°ú ÏßÑÌñâÌïòÏòÄÏúºÎ©∞(Figure 1) κàÏóê ÎåÄÌïú ÏÑ∏ Î∂Ä ÎÇ¥Ïö©ÏùÄ Figure 2ÏôÄ Table 1Ïóê Í∏∞ÏàÝÌïòÏòÄÎã§.
Î∂ÑÏÑùÏóê Ïù¥Ïö©ÎêÝ ÏǨÎûåκà ÏãúΣåÎäî ÏπòÍ≥ºÏö© Ìï∏ÎìúÌîºÏä§Î•º Ïù¥Ïö©Ìïò Ïó¨ 2√ó5 cm ÌŨÍ∏∞ΰú ÏÝàÎã®Ìïú ÌõÑ ÏïàÏ™ΩÍ≥º Î∞îÍπ•Ï™Ω ÌëúΩ¥ÏùÑ ÏïΩ 1 mm ÏÝïÎèÑ Ïó∞ÎßàÌïòÏó¨ ÌëúΩ¥Ïùò ÌÜÝÏñë Î∞è Ïò§ÏóºÎ¨ºÏßàÏùÑ ÏÝúͱ∞ÌïòÏòÄÎã§. κàÏùò ÏÉÅÌÉúÏóê Îî∞Îùº 6% Ï∞®ÏïÑÏóºÏÜåÏÇ∞ÎÇòÌä∏Ε®(sodium hypochlorite, Sigma-Aldrich Inc., St. Louis, MO, USA) ÏàòÏö©Ïï°ÏùÑ 5-10Î∂Ñ Ï≤òζ¨ÌïòÍ≥Ý 3Ï∞® ϶ù ΕòÏàòΰú ÏÑ∏Ï≤ôÌïòÏó¨ ÌëúΩ¥ Ïò§ÏóºÎ¨ºÍ≥º ÌïµÏÇ∞ Ïò§ÏóºÎ¨ºÏùÑ ÏÝúͱ∞ÌïòÏòÄÎã§. ÏÑ∏Ï≤ôÌïú ÏãúΣåÎäî Ψ¥Í∑Ý ÏûëÏóÖÎåÄÏóêÏÑú 24ÏãúÍ∞Ñ Ïù¥ÏÉÅ ÏûêÏó∞ ͱ¥Ï°∞Ìïú ÌõÑ ÏûêÏô∏ÏÑÝÏùÑ 15Î∂ÑÍ∞Ñ Ï°∞ÏǨÌïòÏòÄÏúºÎ©∞ Í∑πÎèôÍ≤∞Î∂ÑÏáÑÍ∏∞(Freezer Mill 6770, SPEX SamplePrep, Metuchen, NJ, USA)Εº ÌܵÌï¥ Î∂ÑÎßê ÏÉÅÌÉúΰú ÏÝúÏ°∞ÌïòÏó¨ Ïã§Ìóò ÏÝÑÍπåÏßÄ 4‚ÑÉÏóêÏÑú Î≥¥Í¥ÄÌïòÏòÄÎã§.
Ïã§Î¶¨Ïπ¥(Silica) Ï∂îÏ∂úÎ≤ïÏùÑ ÌܵÌïú DNA Ï∂îÏ∂úÏùÄ Rohland and Hofreiter(2007)Í∞Ä ÏÝúÏãúÌïú Î∞©Î≤ïÏùÑ ÌôúÏö©ÌïòÏòÄÎã§. ÏǨÎûåκà Î∂ÑÎßê ÏãúΣå ÏïΩ 0.5 gÏóê extraction solution (0.5 M EDTA, 0.25 mg/ml proteinase K, pH 8.0) 10 mlΕº ÎÑ£ÏùÄ ÌõÑ ÏïΩ 16ÏãúÍ∞Ñ ÎèôÏïà ÏÉÅÏò® ÏóêÏÑú ͵êÎ∞òÌïòÏó¨ ÌÉàÏπºÏäòÌôî Î∞òÏùëÏùÑ ÏúÝÎèÑÌïòÏòÄÏúºÎ©∞ ÏùåÏѱÎåÄÏ°∞͵∞ (negative control)ÎèÑ Ìï®Íªò ϧÄÎπÑÌïòÏòÄÎã§. Ïö©Ìï¥Îêú ÏãúΣåÎäî 5,000√ógÏóêÏÑú 2Î∂Ñ ÎèôÏïà ÏõêÏã¨Î∂Ñζ¨ÌïòÍ≥Ý ÏÉÅÏ∏µÏï°ÏùÑ 50 ml ÌäúÎ∏å Ïóê ÏòÆÍ∏¥ ÌõÑ 40 mlÏùò binding buffer (5 M GuSCN, 25 mM NaCl, 50 mM Tris)ÏôÄ 100 ŒºlÏùò Ïã§Î¶¨Ïπ¥ ÌòÑÌÉÅÏï°ÏùÑ Ï≤®Í∞ÄÌïòÏòÄ Îã§. 30% ÏóºÏÇ∞(HCl)ÏùÑ Ïù¥Ïö©ÌïòÏó¨ pH 4.3~4.5ΰú Ï°∞ÏÝàÌïú Îí§ Îπõ ÏùÑ Ï∞®Îã®Ìïú ÏÉÅÌÉúΰú Ïã§Ïò®ÏóêÏÑú 3ÏãúÍ∞Ñ ÎèôÏïà Ï≤òζ¨ÌïòÏòÄÏúºÎ©∞ 5,000√ógÏóêÏÑú 2Î∂ÑÍ∞Ñ ÏõêÏã¨Î∂Ñζ¨ÌïòÏòÄÎã§. Î∂Ñζ¨Îêú ÏÉÅÏ∏µÏï°ÏùÑ ÏÝú ͱ∞Ìïú Îí§ 1 mlÏùò binding bufferΕº Ï≤®Í∞ÄÌïòÏó¨ Ïã§Î¶¨Ïπ¥ ÌéÝζø (pellet)ÏùÑ ÎÖπÏù¥Í≥Ý Îã§Ïãú 16,000√ógÏóêÏÑú 15Ï¥àÍ∞Ñ ÏõêÏã¨Î∂Ñζ¨ÌïòÏó¨ ÏÉÅÏ∏µÏï°ÏùÑ ÏÝúͱ∞ÌïòÏòÄÎã§. Í∑∏Îã§Ïùå washing buffer (50% Ethanol, 125 mM NaCl, 10 mM Tris, 1 mM EDTA, pH 8.0)Εº ÎÑ£Ïñ¥ Îëê Î≤à ÏÑ∏Ï≤ôÌïú ÌõÑ Ïã§Ïò®ÏóêÏÑú 15Î∂ÑÍ∞Ñ ÏûêÏó∞ͱ¥Ï°∞ ÌïòÏòÄÎã§. ͱ¥Ï°∞Îêú Ïã§Î¶¨Ïπ¥Îäî TE buffer 50 ŒºlΕº Ï≤®Í∞ÄÌïòÏó¨ 10Î∂ÑÍ∞Ñ ÍµêÎ∞òÌïú ÌõÑ, 16,000√ógÏóêÏÑú ÏõêÏã¨Î∂Ñζ¨ÌïòÏó¨ DNAΕº Ï∂îÏ∂úÌïòÏòÄÎã§. Ï∂îÏ∂úÎêú DNAÎäî ÏÉàΰúÏö¥ ÌäúÎ∏åÏóê ÏòÆÍ≤® Ïã§Ìóò ÏÝÑÍπåÏßÄ - 40‚ÑÉÏóêÏÑú Î≥¥Í¥Ä ÌïòÏòÄÎã§.
ÌîºÏû•ÏûêÏùò Ι®Í≥Ñ Í≥ÑÌܵÌòï Î∂ÑÏÑùÏùÑ ÏúÑÌï¥ mtDNAÏùò Í≥ºÎ≥ÄÏù¥Î∂Ä ÏúÑ(hyper-variable region; HVR)Εº Î∂ÑÏÑùÌïòÏòÄÎã§. mtDNAΕº ϶ùÌè≠ÌïòÎäîÎç∞ ÏǨÏö©Îêú primerÎäî Îëê Ï¢ÖΕòΰú ϶ùÌè≠ ÏÇ∞ΨºÏùò ÌŨÍ∏∞ Í∞Ä 146~215 bpsÍ∞Ä ÎêòÎèÑΰù ÏÝúÏûëÎêòÏóàÏúºÎ©∞ primerÏùò ÏóºÍ∏∞ÏÑú Ïó¥ÏùÄ Îã§ÏùåÏùò ÎֺΨ∏ÏóêÏÑú ÏÝúÏãúÎêú Í≤ÉÍ≥º Í∞ôÎã§(Jee et al., 2008; Yonsei DNA Profiling Group, 2017). Ï∂îÏ∂úÌïú DNA 5~10 Œºl ÎßàÎ㧠1 U uracil-DNA glycosylase (UDG, UNG; Thermo Fisher Scientific Inc., San Jose, CA, USA)Εº Ï≤òζ¨ ÌõÑ DNA ϧëÌï©Ìö®ÏÜå(AmpliTaq Gold DNA polymerase, Thermo Fisher Scientific Inc., Waltham, MA, USA)Εº Ïù¥Ïö©ÌïòÏó¨ ϧëÌï©Ìö®ÏÜå Ïó∞ÏáÑÎ∞òÏùë(polymerase chain reaction; PCR)ÏùÑ ÏàòÌñâÌïòÏòÄÎã§. AmpliTaq Gold DNA polymerase (Thermo Fisher Scientific Inc., Waltham, MA, USA) 2.5 units, 10 pmol primer pairs 3 Œºl, 2.5 mM dNTPs 3 Œºl, 10X PCR Gold buffer (Thermo Fisher Scientific Inc., Waltham, MA, USA) 3 Œºl, MgCl2 2 Œºl Εº Ï≤®Í∞ÄÌïòÏó¨ ÌòºÌï©Î¨º(mixture)ÏùÑ ÎßåÎìÝ ÌõÑ Ï∂îÏ∂úÌïú DNAΕº ÎÑ£ Í≥Ý 3Ï∞® ϶ùΕòÏàòΕº Ïù¥Ïö©Ìï¥ ÏµúÏ¢Ö 30 Œºlΰú ÎßûÏ∂îÏóàÎã§. ÏúÝÏÝÑÏûê ϶ù Ìè≠Í∏∞(Veriti thermal cycler, Thermo Fisher Scientific Inc., Waltham, MA, USA)Εº ÏǨÏö©ÌïòÏó¨ 94‚ÑÉÏóêÏÑú 30Ï¥à denaturation, 54~62‚ÑÉÏóêÏÑú 30Ï¥à annealing, 72‚ÑÉÏóêÏÑú 1Î∂Ñ extension Í≥º ÏÝïÏùÑ 45Ìöå Î∞òÎ≥µÌïòÏòÄÏúºÎ©∞, 72‚ÑÉÏóêÏÑú 7Î∂ÑÍ∞Ñ last extension Ïãú Ϻ∞Îã§. Yonsei DNA Profiling GroupÏù¥ ÏÝúÏãúÌïú mini-primer setΕº ÏǨÏö©ÌïòÏó¨ PCRÏùÑ ÏàòÌñâÌïòÍ∏∞ ÏúÑÌïú ÌòºÌï©Î¨º Ï°∞ÏѱÏùÄ ÏúÑÏôÄ Í∞ôÏúºÎÇò 3Ï∞® ϶ùΕòÏàòΕº Ïù¥Ïö©Ìï¥ ÏµúÏ¢Ö 25 ŒºlÏúºÎ°ú ÎßûÏ∂îÏóàÏúºÎ©∞, ÏúÝÏÝÑÏûê ϶ùÌè≠Í∏∞Εº ÏǨÏö©ÌïòÏó¨ 95‚ÑÉÏóêÏÑú 11Î∂Ñ initial denaturation ÌõÑ, 95‚ÑÉÏóêÏÑú 20Ï¥à denaturation, 50‚ÑÉÏóêÏÑú 20Ï¥à annealing, 72‚ÑÉÏóêÏÑú 30Ï¥à extension Í≥ºÏÝïÏùÑ 42Ìöå Î∞òÎ≥µÌïòÏòÄÏúºÎ©∞, 72‚ÑÉ ÏóêÏÑú 7Î∂ÑÍ∞Ñ last extension ÏãúϺ∞Îã§. PCR Í≥ºÏÝïÏùÑ ÎßàÏπú ϶ùÌè≠ ÏÇ∞ΨºÏùÄ ÏûêÎèôÏÝÑÍ∏∞ÏòÅÎèôÏû•Ïπò(HDA-GT12, eGENE Inc., Irvine, CA, USA)Εº ÌܵÌï¥ ÌôïÏù∏ÌïòÏòÄÎã§. ÏûêÎèôÏÝÑÍ∏∞ÏòÅÎèôÏû•ÏπòΕº ÌܵÌï¥ ÌôïÏù∏Ìïú ϶ùÌè≠ ÏÇ∞ΨºÏùÄ Exo-SAP-IT (Thermo Fisher Scientific Inc., Waltham, MA, USA)ÏúºÎ°ú ÏÝïÏÝúÌïú Îã§Ïùå, ÏûêÎèôÏóºÍ∏∞ÏÑúÏó¥ Î∂ÑÏÑùÏû•Ïπò(Prism 3100, Thermo Fisher Scientific Inc., Waltham, MA, USA)ΰú ÏóºÍ∏∞ÏÑúÏó¥ÏùÑ Î∂ÑÏÑùÌïòÏòÄÎã§.
PCR ϶ùÌè≠ ÏÇ∞ΨºÏùÄ Qiaquick PCR purification kit (Qiagen, Hilden, Germany)Εº ÏǨÏö©ÌïòÏó¨ ÏÝïÏÝúÌïú Îí§, pGEM-T Easy vector (Promega, Madison, WI, USA)Ïóê ÌťΰúÎãùÌïòÏòÄÎã§. Ligation mixtureÎäî heat shock Î∞©Î≤ïÏóê ÏùòÌï¥ E. Coli DH5Œ± competent cellΰú ÌòïÏßàÏÝÑÌôòÎêòÏóàÎã§. TransformantsÎäî ampicillin, IPTGÏôÄ X-galÏù¥ Ï≤®Í∞ÄÎêú Luria-Bertani (LB) Î∞∞ÏßÄÏóêÏÑú ÌôîÏù¥Ìä∏ Îã®Ïùº colony 10-20Í∞úΕº ÏÑÝÎ≥ÑÌïòÏó¨ LB Ïï°Ï≤¥ Î∞∞ÏßÄÏóêÏÑú 12-14ÏãúÍ∞Ñ Î∞∞ÏñëÌïòÍ≥Ý Qiaprep Spin Miniprep kit (Qiagen, Hilden, Germany)Εº ÏǨÏö©ÌïòÏó¨ ÌîåÎùºÏä§ÎØ∏Îìú DNAΕº Ï∂îÏ∂úÌïòÏó¨ ÏûêÎèô ÏóºÍ∏∞ÏÑúÏó¥Î∂ÑÏÑùÏû•Ïπò(Prism 3100, Thermo Fisher Scientific Inc., Waltham, MA, USA)ΰú ÏóºÍ∏∞ÏÑúÏó¥ÏùÑ Î∂ÑÏÑùÌïòÏòÄÎã§.
피장자의 mtDNA 하플로그룹 분석은 pGEM-T Easy vector에 삽입된 gene의 염기서열분석 결과와 두 종류의 primer set를 이용하여 증폭한 direct PCR 염기서열 분석 결 과를 교차대조하였다.
mtDNAÏùò Î≥ÄÏù¥Ìòï(haplotype)ÏùÄ CLC Main Workbench 6 ÌîÑΰúÍ∑∏Îû®(QIAGEN Bioinformatics, Aarhus, Middle Jutland, Denmark)ÏùÑ Ïù¥Ïö©ÌïòÏó¨ ÌëúϧÄÏóºÍ∏∞ÏÑúÏó¥(revised Cambridge Reference Sequence; rCRS) (Andrews et al., 1999)ÏùÑ Í∏∞준 ÏúºÎ°ú Í≤∞ÏÝïÎêòÏóàÏúºÎ©∞ ÏãúΣåÏùò mtDNA Î≥ÄÏù¥ÌòïÏùÑ ÌÜÝÎåÄΰú mtDNA manager (http://mtmanager.yonsei.ac.kr, Yonsei University College of Medicine, Seoul, Korea)ÏôÄ HaploGrep 2 (https:// haplogrep.uibk.ac.at, Division of Genetic Epidemiology, Medical University of Innsbruck, Innsbruck, Austria)ÏóêÏÑú ÌïòÌîåΰúÍ∑∏ΣπÏùÑ ÎèôÏÝïÌïòÏó¨ Ïù¥Ï§ëÏúºÎ°ú ÎπÑ͵ê Î∂ÑΕòÌïòÏòÄÎã§.(Lee et al., 2008b; Weissensteiner et al., 2016).
Î∂ÄÏÇ∞ ÌôîΙÖÎèô Î∂ÑΨò͵∞ÏóêÏÑú Ï∂úÌÜÝÎêú ÏòõÏǨÎûå κà 8Í∞úÏ≤¥Î•º ÎåÄ ÏÉÅÏúºÎ°ú Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Ïã§ÌóòÏùÑ ÏßÑÌñâÌïòÏòÄÏúºÎ©∞ ÌëúϧÄÏóºÍ∏∞ÏÑúÏó¥ Í≥ºÏùò ÎπÑ͵êΕº ÌܵÌï¥ mtDNA Í≥ºÎ≥ÄÏù¥Î∂ÄÏúÑ(HVR I, HVR II, HVR III)Εº Î∂ÑÏÑùÌïòÍ≥Ý Í∞úÏ≤¥Î≥Ñ ÌïòÌîåΰúÌÉÄÏûÖÍ≥º ÌïòÌîåΰúÍ∑∏ΣπÏùÑ ÌôïÏù∏ÌïòÏòÄÎã§. Ïù¥ ϧë 3Í∞úÏ≤¥Îäî ÌïòÌîåΰúÌÉÄÏûÖ Î∂ÑÏÑùÏù¥ Í∞ÄÎä•ÌïòÏòÄÍ≥Ý mtDNA managerÏôÄ HaploGrep 2 ÌîÑΰúÍ∑∏Îû®ÏùÑ Ïù¥Ïö©ÌïòÏó¨ Ìïò ÌîåΰúÍ∑∏ΣπÏùÑ Í≤∞ÏÝïÌïòÏòÄÎã§. T vectorÏóê ÏÇΩÏûÖÌïú target insertÏùò ÏóºÍ∏∞ÏÑúÏó¥Í≥º direct PCR ÏóºÍ∏∞ÏÑúÏó¥ÏùÑ ÍµêÏ∞®ÎåÄÏ°∞ÌïòÏó¨ ÌôïÏù∏Îêú ÎèôÏùºÌïú Í≤∞Í≥ºÎäî Table 2ÏôÄ Í∞ôÎã§.
Determined haplotype and haplogroup for each variants
mtDNA manager ÌîÑΰúÍ∑∏Îû®ÏóêÏÑú 11Ìò∏Ψò ÌîºÏû•Ïûê(BH-1)Îäî A5a Í∑∏Σπ, 21Ìò∏Ψò ÌîºÏû•Ïûê(BH-5)Îäî D4a Í∑∏Σπ, 26Ìò∏Ψò ÌîºÏû• Ïûê(BH-8)Îäî M*-489 Í∑∏ΣπÏúºÎ°ú ÎèôÏÝïÎêòÏñ¥ ÌïòÌîåΰúÍ∑∏ΣπÏù¥ A Í∑∏Σπ 1Í∞úÏ≤¥, D Í∑∏Σπ 1Í∞úÏ≤¥, M Í∑∏Σπ 1Í∞úÏ≤¥Î°ú Î∂ÑÏÑùÎêòÏóàÎã§. HaploGrep 2 ÌîÑΰúÍ∑∏Îû®ÏóêÏÑúÎäî 11Ìò∏Ψò ÌîºÏû•ÏûêÍ∞Ä A5a Í∑∏Σπ, 21Ìò∏Ψò ÌîºÏû•ÏûêÎäî D4a Í∑∏Σπ, 26Ìò∏Ψò ÌîºÏû•ÏûêÎäî M4"67+16311 Í∑∏ΣπÏù¥ Í∞ÄÏû• ÎÜíÏùÄ qualityΰú ÎèôÏÝïÎêòÏñ¥ A Í∑∏ΣπÏù¥ 1Í∞úÏ≤¥, D Í∑∏ΣπÏù¥ 1Í∞úÏ≤¥, M Í∑∏ΣπÏù¥ 1Í∞úÏ≤¥Î°ú ÎèôÏÝïÎêòÏóàÎã§. Ïù¥Îäî mtDNA manager ÌîÑΰúÍ∑∏Îû®ÏóêÏÑú ÌôïÏù∏Ìïú ÌïòÌîåΰúÍ∑∏Σπ Î∂ÑÏÑù Í≤∞Í≥ºÏôÄ Îèô ÏùºÌïòÏòÄÏúºÎ©∞, Ïù¥ ϧë 11Ìò∏Ψò ÌîºÏû•ÏûêÏùò A5a Í∑∏ΣπÍ≥º 26Ìò∏Ψò Ìîº Ïû•ÏûêÏùò M4"67+16311 Í∑∏ΣπÏùÄ 100% qualityΰú ÎèôÏÝïÎêòÏñ¥ Í≤∞ Í≥ºÏùò ÏãÝ΢∞ÎèÑΕº ÎÜíÏó¨Ï§ÄÎã§. Îã§Îßå, ÎèôÏùºÌïú mtDNA Î≥ÄÏù¥Ìòï Î∂Ñ ÏÑù Í≤∞Í≥ºÎ•º Ïù¥Ïö©ÌïòÏó¨ ÎπÑ͵êÌïòÏòÄÏùåÏóêÎèÑ Î∂à͵¨ÌïòÍ≥Ý 26Ìò∏Ψò Ìîº Ïû•ÏûêÏùò Í≤∞Í≥ºÏ≤òÎüº ÌîÑΰúÍ∑∏Îû®Î≥Ñΰú ÏÑ∏Î∂Ä ÌïòÌîåΰúÍ∑∏ΣπÏù¥ Îã§Î•¥Í≤å ÎèôÏÝïÎêòÎäî Í≤ÉÏùÄ Í∞Å ÌîÑΰúÍ∑∏Îû®ÏóêÏÑú ÌôúÏö©ÎêòÎäî databaseÏùò Ï∞®Ïù¥ ÏôÄ coding regionsÏóê ÎåÄÌïú Î∂ÑÏÑù Í≤∞Í≥ºÏùò ÎàÑÎùΩ Îì±Ïóê ÏùòÌïú Í≤ÉÏúº ΰú ÏǨΣåÎêúÎã§.
11Ìò∏Ψò ÌîºÏû•ÏûêÍ∞Ä ÏÜçÌïòÎäî ÌïòÌîåΰúÍ∑∏Σπ A5aÎäî A Í∑∏ΣπÏóê ÏÜçÌïòΩ∞ N Í∑∏ΣπÏúºÎ°úÎ∂ÄÌÑ∞ ÌååÏÉùÎêòÏóàÎã§. A Í∑∏ΣπÏùÄ Ìïú͵≠Ïù∏Ïùò ÏïΩ 7-15%Í∞Ä ÏÜçÌï¥ÏûàÏúºÎ©∞ Ïù¥ ϧë A5 Í∑∏ΣπÏùÄ Ïö∞ζ¨ÎÇòÎùºÏôÄ ÏùºÎ≥∏, 몽 Í≥®, Î≤ÝÌä∏ÎÇ®ÏóêÏÑú ÏÝúÌïúÏÝÅÏúºÎ°ú ÎÇòÌÉÄÎÇúÎã§(Kong et al., 2003; Maruyama et al., 2003; Tanaka et al., 2004; Derenko et al., 2007; Jin et al., 2009). 21Ìò∏Ψò ÌîºÏû•ÏûêÎäî ÌïòÌîåΰúÍ∑∏Σπ D4a Í∑∏ΣπÏúºÎ°ú Î∂ÑÏÑùÎêòÏóàÎäîÎç∞ D4 Í∑∏ΣπÏùÄ Ïö∞ζ¨ÎÇòÎùºÏôÄ ÏùºÎ≥∏, ϧë͵≠, ΙΩÍ≥®ÏóêÏÑú 20~30%Ïùò ÎÜíÏùÄ ÎπÑÏú®Î°ú Î∞úÍ≤¨ÎêòÎäî Í∑∏ΣπÏù¥Îã§(Kim et al., 2015). ÌïòÌîåΰúÍ∑∏Σπ D4Îäî DÏùò ÌïòÏúÑÍ∑∏ΣπÏúºÎ°ú Ïö∞ζ¨ÎÇò ÎùºÎ•º ÎπÑΰØÌï¥ ÏùºÎ≥∏, ϧë͵≠ Î∂ÅÎ∂Ä ÏǨÎûåÎì§Í≥º Í∞ôÏùÄ ÎèôÎ∂ÅÏïÑÏãúÏïÑ ÌòÑ ÎåÄÏù∏Îì§ÏóêÍ≤å ÎߧÏö∞ ÎÜíÏùÄ ÎπàÎèÑΰú Ï∂úÌòÑÌïòÎäî ÎåÄÌëúÏÝÅÏù∏ ÎèôÎ∂ÅÏïÑÏãú ÏïÑÏùò ÎØ∏ÌÜÝÏΩòÎìúζ¨ÏïÑ Î≥ÄÏù¥ÌòïÏù¥Îã§(Tanaka et al., 2004; Lee et al., 2006). ÌïòÌîåΰúÍ∑∏Σπ DÎäî M Í∑∏ΣπÏúºÎ°úÎ∂ÄÌÑ∞ Î∂ÑÍ∏∞Îêú Í∑∏Σπ ÏúºÎ°ú ÌòÑÎåÄ Ìïú͵≠Ïù∏Ïùò ÏïΩ 32.4%Í∞Ä ÌïòÌîåΰúÍ∑∏Σπ Dΰú ÎÇòÌÉÄÎÇúÎ㧠(Jin et al., 2009). 26Ìò∏Ψò ÌîºÏû•ÏûêÏùò ÌïòÌîåΰúÍ∑∏Σπ M4"67Ïù¥ ÏÜçÌïú M Í∑∏ΣπÏùÄ ÏïÑÏãúÏïÑÏóêÏÑú Í∞ÄÏû• ÏùºÎ∞òÏÝÅÏù∏ ÌïòÌîåΰúÍ∑∏ΣπÏúºÎ°ú Ïö∞ζ¨ÎÇòÎùºÏóêÏÑú ÏïΩ 60% ÎπàÎèÑΕº Î≥¥Ïù¥Îäî Í∑∏ΣπÏù¥Îã§(Hong et al., 2014).
ÏúÝÏÝÅÏßÄÏóêÏÑú Ï∂úÌÜÝÎêòÎäî Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÎäî ÏÑÝÏǨÏãúÎåÄÎÇò ÏǺ ͵≠ÏãúÎåÄ ÏǨÎûåκàÏóê ÎπÑÌï¥ ÏãúÎåÄÏÝÅ, Ïó≠ÏǨÏÝÅ Í∞ÄÏπòÎÇò Í≥ÝÍ≥ÝÌïôÏÝÅ Ïùò ÏùòÍ∞Ä Í∑∏ζ¨ ÎÜíÎã§Í≥Ý ÌïÝ Ïàò ÏóÜÎã§. ÌïòÏßÄÎßå Î∂ÑΨò͵∞ÏóêÏÑú ÎåÄÎüâÏúºÎ°ú Ï∂úÌÜÝÎêòÎäî ÏǨΰÄÍ∞Ä ÎßéÏùÄ ÎßåÌź ÏßëÎã® ÎÇ¥Ïùò ÏúÝÏÝÑÏÝÅ Î≥ÄÏù¥ÌòïÏùò Î≥Ä ÌôîÎÇò Î∂ÑÌè¨, ÎπàÎèÑÏùò ÏñëÏÉÅÏùÑ ÌܵÍ≥ÑÌïôÏÝÅÏúºÎ°ú Î∂ÑÏÑùÌïòÏó¨ ÏßëÎã®Ïùò Ïù¥ÎèôÏùÑ ÏѧΙÖÌïòÍ∏∞Ïóê ÏÝÅÌï©ÌïòÎã§. ÎòêÌïú ÏãúÎåÄÎ≥Ñ Ïù∏ΕòÏúÝÏÝÑÌïôÏÝÅ ÌäπÏѱÏùÑ Ïó∞͵¨ÌïòÍ∏∞ ÏúÑÌïú ÎåÄÏÉÅÏúºÎ°ú ÎߧÏö∞ ÏúÝÏö©ÌïòΩ∞, ÌòÑÎåÄ ÌïúÎ∞ò ÎèÑ ÌòÑÎåÄÏù∏ ÏßëÎã®Ïùò ÌòïÏѱÍ≥º ÏßëÎã®ÏúÝÏÝÑÌïôÏÝÅ Î≥ÄÌôî ÏñëÏÉÅÏùÑ Íµ¨Ï≤¥ ÏÝÅÏúºÎ°ú ÌååÏïÖÌïÝ Ïàò ÏûàÎã§ÎäîÎç∞ ϧëÏöîÌïú Í∞ÄÏπòÍ∞Ä ÏûàÎã§.
Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÎäî ÏÝÑ͵≠ÏóêÏÑú Ï∂úÌÜÝÎêòÍ≥Ý ÏûàÎäî ÎßåÌź ÏßÄÏó≠ Í∞Ñ ÌïòÌîåΰúÍ∑∏Σπ Î∂ÑÌè¨Ïùò ÎπÑ͵êÍ∞Ä Í∞ÄÎä•ÌïòÎã§. Hong et al. (2015) Ïóê ÏùòÌïòΩ¥, ͵≠ÎÇ¥ 7Í∞ú ÏßÄÏó≠ ÌòÑÎåÄÏù∏Ïùò mtDNA ÌïòÌîåΰúÍ∑∏Σπ Î∂Ñ ÏÑùÏùÑ ÌܵÌïú ÏúÝÏÝÑÏÝŠͱ∞ζ¨Î•º ÎπÑ͵êÌïú Í≤∞Í≥º, ÏßÄÏó≠ Í∞ÑÏóêÎäî ÌäπÎ≥ÑÌïú Ï∞®Ïù¥Î•º Î≥¥Ïù¥ÏßÄ ÏïäÏúºÎÇò ÏÝúÏ£ºÎèÑÏôÄÎäî Ï∞®Ïù¥Í∞Ä ÏûàÎäî Í≤ÉÏúºÎ°ú Î∞ù ÌòÄÏßÑ Î∞î ÏûàÎã§. ÏòõÏǨÎûå κàÏùò Î∂ÑÏûêÏúÝÏÝÑÌïô Ïó∞͵¨Îäî ÏùºÎ∂Ä ÏãúΣåÎßå ÏúºÎ°ú Í≥ÑÌܵÏùÑ ÎÖºÌïÝ ÏàòÎäî ÏóÜÏßÄÎßå ÎèÑÏ∂úÎêú DNA ÏóºÍ∏∞ÏÑúÏó¥ ÎπÑ͵ê Εº ÌܵÌï¥ Í≥ºÍ±∞Ïóê ÎÇòÌÉÄÎÇòÏßÄ ÏïäÏïòÎçò Î≥ÄÏù¥ÌòïÍ≥ºÏùò ÎπÑ͵êÍ∞Ä Í∞ÄÎä• ÌïòÎã§. ϶â, Í≥ºÍ±∞ÏóêÏÑúÎ∂ÄÌÑ∞ ÌòÑÎåÄÏù∏Ïóê Ïù¥Î•¥Í∏∞ÍπåÏßÄ ÏúÝÏÝÑÏÝÅ Î≥ÄÏù¥ ÌòïÏùò Î≥ÄÌôîΕº Î∂ÑÏÑùÌïòÍ≥Ý Ïù¥Î•º ÌÜÝÎåÄΰú ÌïòÌîåΰúÍ∑∏Σπ Î∂ÑÌè¨ÎèÑÏôÄ ÎπàÎèÑ Îì±ÏùÑ ÎπÑ͵êÎ∂ÑÏÑù Ìï¥ Î≥º Ïàò ÏûàÎã§.
ÏòõÏǨÎûå κà ÏãúΣåÎäî Ïò§Îû´ÎèôÏïà ÎïÖÏÜçÏóê ÎߧÏû•ÎêòÏñ¥ ÏûàÏóàÍ∏∞ Îïå Ψ∏Ïóê DNAÍ∞Ä ÏÝàÌé∏ÌôîÎêòÎäî ÌäπÏѱÏùÑ Í≥ÝÎݧÌïòÏó¨ ϶ùÌè≠ ÏÇ∞ΨºÏùò ÌŨ Í∏∞Í∞Ä 146~215 bpsΰú ϶ùÌè≠ÎêòÎäî primerΕº ÏǨÏö©ÌïòÏòÄÍ≥Ý ÏãúΣå Ïùò mtDNA HVR I (16024~16383), HVR II (57~372), HVR III (438~574)ÏóêÏÑú ÎÇòÌÉÄÎÇòÎäî Î≥ÄÏù¥ÌòïÏùÑ Î∂ÑÏÑùÌïòÍ∏∞ ÏúÑÌïòÏó¨ 9 Í∞ú Î∂ÄÏúÑΰú ÎÇòÎàÑÏñ¥ Ïã§ÌóòÏùÑ ÏßÑÌñâÌïòÏòÄÎã§. Îߧ Ìöå Ï∂îÏ∂úÍ≥º ϶ùÌè≠ Í≥ºÏÝï Ïãú ÏùåÏѱÎåÄÏ°∞͵∞ÏùÑ Ìï®Íªò Ïã§ÌóòÌï®ÏúºÎ°úÏç® Ïã§Ìóò Í≥ºÏÝï ϧëÏóê Î∞úÏÉùÌïÝ Ïàò ÏûàÎäî DNA Ïò§Ïóº Ïó¨Î∂ÄΕº ÌôïÏù∏ÌïòÏòÄÍ≥Ý UDGΕº Ï≤ò ζ¨Ìïú DNAÏóê Îëê Ï¢ÖΕòÏùò primer setΕº ÏǨÏö©ÌïòÏó¨ ϶ùÌè≠Ìïú direct PCR ÏóºÍ∏∞ÏÑúÏó¥Í≥º clone ÏóºÍ∏∞ÏÑúÏó¥ÏùÑ ÍµêÏ∞®ÎåÄÏ°∞ÌïòÏó¨ Îèô ÏùºÌïú Í≤∞Í≥ºÎßåÏùÑ ÎèÑÏ∂úÌï®ÏúºÎ°úÏç® Í≤∞Í≥ºÏùò ÏãÝ΢∞ÎèÑΕº ÎÜíÏòÄÎã§. Îòê Ìïú ÌôîΙÖÎèô ÏúÝÏÝÅÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκàÏùò Î∂ÑÏÑù Í≤∞Í≥ºÎ•º ÏÝÑÏ≤òζ¨ ÏôÄ Î∂ÑÏÑùÏóê Ï∞∏Ïó¨Ìïú Ïó∞͵¨ÏõêÎì§Ïùò mtDNA ÌïòÌîåΰúÌÉÄÏûÖÍ≥º ÎπÑ͵ê ÌïòÏó¨ Ïó∞͵¨ÏûêÏóê ÏùòÌïú Ïã§Ìóò Í≤∞Í≥ºÏùò Ïò§Ïóº Í∞ÄÎä•ÏѱÏùÑ Î∞∞ÏÝúÌïòÏòÄ Îã§(Table 3).
The results of contamination candidates for determined mtDNA variants and haplogroup analysis
Î∂ÄÏÇ∞ ÌôîΙÖÎèô ÏúÝÏÝÅÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκàÏùò mtDNA ÌïòÌîåΰú Í∑∏Σπ Î∂ÑÏÑù Í≤∞Í≥º mtDNA managerÏôÄ HaploGrep 2 ÌîÑΰúÍ∑∏Îû® ÏóêÏÑú 3Í∞úÏ≤¥Ïùò ÌïòÌîåΰúÍ∑∏ΣπÏù¥ ÎèôÏÝïÎêòÏóàÎäîÎç∞ Ïù¥ ϧë 26Ìò∏Ψò Ìîº Ïû•ÏûêÏùò ÏÑ∏Î∂Ä ÌïòÌîåΰúÍ∑∏ΣπÏùÄ ÌôúÏö©ÎêòÎäî databaseÏùò Ï∞®Ïù¥, coding regionsÏóê ÎåÄÌïú Î∂ÑÏÑù Í≤∞Í≥ºÏùò ÎàÑÎùΩ Îì±ÏúºÎ°ú Ïù∏Ìï¥ ÌïòÌîåΰúÍ∑∏Σπ Í≤∞ÏÝï Ïãú Ï∞®Ïù¥Í∞Ä ÎÇòÌÉÄÎÇú Í≤ÉÏúºÎ°ú ÌåêÎã®ÎêúÎã§. ÌäπÌûà HaploGrep 2 ÌîÑΰúÍ∑∏Îû®Ïùò Í≤ΩÏö∞ ÎπÑÍ≤∞ÏÝï͵¨Í∞ÑÏùÑ ÏÝúÏô∏Ìïú Î≤îÏúÑΕº Î≥ÑÎèÑΰú Ïѧ ÏÝïÌïòÏó¨ ÌïòÌîåΰúÍ∑∏ΣπÏùÑ Í≤∞ÏÝïÌïÝ Ïàò ÏûàÏúºÎØÄΰú Îã§ÌòïÏѱ ÎàÑÎùΩÏúºÎ°ú Ïù∏Ìïú Ïò§Î•òΕº ϧÑÏùº Ïàò ÏûàÏóàÎã§. ÎèôÏÝïÎêú ÌïòÌîåΰúÍ∑∏Σπ Í∞ÄÏö¥Îç∞ D4 Í∑∏ΣπÏùÄ ÎåÄÌëúÏÝÅÏù∏ ÎèôÎ∂ÅÏïÑÏãúÏïÑ ÎØ∏ÌÜÝÏΩòÎìúζ¨ÏïÑ Î≥ÄÏù¥ÌòïÏúº ΰú Ìïú͵≠Ïù∏Ïùò 23.8%Í∞Ä ÏÜçÌïòÎäî Í≤ÉÏúºÎ°ú ÏïåÎݧÏÝ∏ ÏûàÏúºÎ©∞ A5a Í∑∏ΣπÏùÄ Ìïú͵≠ÏóêÏÑú ÌäπÏßïÏÝÅÏúºÎ°ú ÎÇòÌÉÄÎÇòÏßÄÎßå Í∑πÌûà ÎÇÆÏùÄ ÎπàÎèÑΰú Î∂ÑÌè¨ÌïòÎäî ÌïòÌîåΰúÍ∑∏ΣπÏù¥Îã§(Jin et al., 2009). ÏòÅÎÇ®ÏßÄÏó≠ ÌòÑÎåÄ Ïù∏Ïùò 32.4%(36/111)Í∞Ä D4 Í∑∏ΣπÏóê ÏÜçÌïòÎäî Í≤ÉÏúºÎ°ú ÎÇòÌÉÄÎǨÎäî Îç∞ Î∂ÄÏÇ∞ ÌôîΙÖÎèôÏóêÏÑú Î∂ÑÏÑùÎêú ÏǨÎûåκà 3Í∞úÏ≤¥ ϧë 1Í∞úÏ≤¥Í∞Ä D4 Í∑∏ΣπÏóê ÏÜçÌïòÎäî Í≤ÉÏúºÎ°ú ÎÇòÌÉÄÎÇú Í≤ÉÏùÄ Î¨¥ÏùòÎØ∏ÌïòÏßÄ ÏïäÎã§(Hong, 2014). ÎøêÎßå ÏïÑÎãàÎùº Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκà Í≤∞Í≥ºÎ•º ÎπÑ͵êÌñàÏùÑ Îïå ÍπÄÌè¨ Ïû•Í∏∞Îèô(2/6), ÍπÄÌè¨ ÌïôÏö¥Î¶¨(11/25), ÏïÑÏÇ∞ ΙÖÏïîζ¨(7/27) ÏóêÏÑúÎèÑ ÌïòÌîåΰúÍ∑∏Σπ DÍ∞Ä ÎÜíÏùÄ ÎπÑÏú®Î°ú ÎÇòÌÉÄÎÇòÎäî Í≤ÉÏùÑ ÌôïÏù∏ÌïÝ Ïàò ÏûàÎã§(Seo et al., 2014; Jin et al., 2015; Kim et al., 2015). Î∞ÄÏßëÎêòÏñ¥ ÏûàÎçò ÏßëÎã®Î¨òÏùò ÌäπÏßïÏùÑ Í≥ÝÎݧÌïòÏó¨ Ïù∏ÏÝëÌïú Í≥≥Ïóê Îߧ Ïû•Îêú ÌîºÏû•Ïûê Í∞ÑÏùò ÎØ∏ÌÜÝÏΩòÎìúζ¨ÏïÑ Î≥ÄÏù¥Ìòï ÎπÑ͵êΕº ÌܵÌï¥ Î™®Í≥Ñ ÏúÝÏó∞Í¥ÄÍ≥ÑΕº Ï∞æÏïÑÎ≥¥Í≥ÝÏûê ÌïòÏòÄÏúºÎÇò Î∂ÑÏÑùÎêú Í∞úÏ≤¥ ÏàòÍ∞Ä ÏÝÅÍ≥Ý ÌîºÏû•Ïûê Í∞ÑÏóê ÌäπÏßïÏù¥ ÎêÝ Ïàò ÏûàÎäî ÌïòÌîåΰúÌÉÄÏûÖÏùÑ Í≥µÏúÝÌïòÍ≥Ý Ïûà ÏßÄ ÏïäÏúºÎØÄΰú Í∞úÏ≤¥ Í∞ÑÏùò Ïó∞Í¥ÄÏѱÏùÑ Ï∞æÏùÑ Ïàò ÏóÜÏóàÎã§. Îî∞ÎùºÏÑú 11Ìò∏ΨòÏôÄ 21Ìò∏Ψò, 26Ìò∏Ψò ÌîºÏû•Ïûê Í∞ÑÏóê Ι®Í≥Ñ ÏúÝÏó∞Í¥ÄÍ≥ÑÎäî ÏóÜ Îäî Í≤ÉÏúºÎ°ú ÌåêÎã®ÎêúÎã§.
Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Î∂ÑÏÑù Í≤∞Í≥º Î∂ÄÏÇ∞ ÌôîΙÖÎèô ÏúÝÏÝÅÏùÄ Î∂ÑΨòÍ∞ÑÏùò ͱ∞ζ¨Í∞Ä ÎߧÏö∞ Í∞ÄÍπåÏõÄÏóêÎèÑ Î∂à͵¨ÌïòÍ≥Ý ÌäπÎ≥ÑÌïú Í∞ÄÏ°±Í¥ÄÍ≥ÑÎèÑ ÏóÜÎäî Îã®ÏàúÌïú Í≥µÎèôΨòÏßÄÏùº Í∞ÄÎä•ÏѱÏùÑ ÏÝúÏãúÌïÝ Ïàò ÏûàÎã§. ÎòêÌïú Î≥∏ Ïó∞͵¨ Îäî ÏòÅÎÇ®ÏßÄÏó≠ÏóêÏÑú Ï∂úÌÜÝÎêú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàΕº ÎåÄÏÉÅÏúºÎ°ú ÏàòÌñâ Ìïú Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Î∂ÑÏÑù Í≤∞Í≥ºÎ°ú ÏÝÅÏùÄ Í∞úÏ≤¥ Ïàòΰú ÌïúÏÝïÎêú Î∂ÑÏÑù ÎßåÏùÑ ÏàòÌñâÌïòÏòÄÍ∏∞ ÎïåΨ∏Ïóê ÏßëÎã®ÏúÝÏÝÑÌïôÏÝÅ ÌäπÏßïÏùÑ ÌååÏïÖÌïòÍ∏∞Îäî Ïñ¥ÎݵÎã§. Í∑∏Îü¨ÎÇò Ïù¥Î≤à Ïó∞͵¨Î•º ÌÜÝÎåÄΰú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÏùò ÏßÄ Ïó≠Î≥Ñ Î∂ÑÏÑù ÏûêΣåÍ∞Ä Ï∂îÍ∞Äΰú ÌôïÎ≥¥ÎêúÎã§Î©¥ mtDNA ÌïòÌîåΰúÌÉÄÏûÖ databaseΕº Ïù¥Ïö©Ìïú ÏúÝÏÝÑÌïôÏÝŠͱ∞ζ¨, ÌïòÌîåΰúÍ∑∏Σπ Î∂ÑÌè¨ÎèÑ, Îπà ÎèÑÏùò Î≥ÄÌôîΕº ÌååÏïÖÌïÝ Ïàò ÏûàÏúºÎ©∞ ÎÇòÏïÑÍ∞Ä ÌòÑÎåÄ Ìïú͵≠Ïù∏Í≥ºÏùò ÎπÑ ÍµêΕº ÌܵÌï¥ Ìïú͵≠Ïù∏Ïùò ÏúÝÏÝÑÌïôÏÝÅ ÏßëÎ㮠͵¨Ï°∞Ïùò Î≥ÄÌôîΕº Ïù¥Ìï¥Ìïò ÎäîÎç∞ ÏúÝÏö©Ìïú ÏûêΣåÍ∞Ä ÎêÝ Ïàò ÏûàÏùÑ Í≤ÉÏúºÎ°ú ÏǨΣåÎêúÎã§.
Î≥∏ Ïó∞͵¨Îäî Î∂ÄÏÇ∞ ÌôîΙÖÎèô Ï°∞ÏÑÝÏãúÎåÄ Î∂ÑΨò͵∞ÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨ ÎûåκàÏùò Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Î∂ÑÏÑùÏùÑ ÌܵÌï¥ Î™®Í≥Ñ Í≥ÑÌܵÌòïÏùÑ ÌååÏïÖÌïòÏòÄ Í≥Ý ÌîºÏû•Ïûê Í∞ÑÏùò Ι®Í≥Ñ ÏúÝÏó∞Í¥ÄÍ≥Ñ Ïó¨Î∂ÄΕº ÌôïÏù∏ÌïòÏòÄÎã§. ÏǨÎûåκà Ïùò DNAÎäî Ïã§Î¶¨Ïπ¥ Ï∂îÏ∂úÎ≤ïÏùÑ ÏǨÏö©ÌïòÏó¨ Ï∂îÏ∂úÌïòÏòÄÏúºÎ©∞ mtDNA Î∂ÑÏÑù Í≤∞Í≥º 3Í∞úÏ≤¥ ÌîºÏû•ÏûêÏùò ÌïòÌîåΰúÌÉÄÏûÖÏù¥ ÌôïÏù∏ÎêòÏóà Îã§. mtDNA managerÏôÄ HaploGrep 2 ÌîÑΰúÍ∑∏Îû®ÏùÑ ÌܵÌïú ÌïòÌîå ΰúÍ∑∏Σπ ÎèôÏÝï Í≤∞Í≥º, 11Ìò∏ΨòÏôÄ 21Ìò∏Ψò ÌîºÏû•ÏûêÎäî A5a, D4a Í∑∏ ΣπÏúºÎ°ú Î∂ÑΕòÎêòÏóàÏúºÎ©∞, 26Ìò∏Ψò ÌîºÏû•ÏûêÎäî M*-489(mtDNA manager ÌîÑΰúÍ∑∏Îû®), M4"67+16311(HaploGrep 2 ÌîÑΰúÍ∑∏Îû®) Í∑∏ΣπÏúºÎ°ú Î∂ÑΕòÎêòÏóàÎã§. ÌîºÏû•Ïûê Î≥Ñ ÌïòÌîåΰúÌÉÄÏûÖÏùÑ ÎπÑ͵êÌñàÏùÑ Îïå Í∞ôÏùÄ Î≥ÄÏù¥ÌòïÏùÑ Í≥µÏúÝÌïòÏßÄ ÏïäÏúºÎØÄΰú ÌîºÏû•Ïûê Í∞ÑÏóê Ι®Í≥Ñ Ïπú Ïó∞Í¥ÄÍ≥ÑÎäî ÏóÜÎäî Í≤ÉÏúºÎ°ú ÌåêÎã®ÎêúÎã§. Ïù¥Î≤à Ïó∞͵¨ Í≤∞Í≥ºÎ•º ÌܵÌï¥ Í≥º ͱ∞ ÌïúÎ∞òÎèÑÏóê ÏÇ¥ÏïòÎçò ÏòõÏǨÎûåÎì§Í≥º ÌòÑÎåÄÏù∏Îì§ Í∞ÑÏùò ÏúÝÏÝÑÌïôÏÝŠͥÄÍ≥ÑΕº Ïù¥Ìï¥ÌïòÍ∏∞ ÏúÑÌïú Í∏∞Ï¥à ÏûêΣåΰú ÌôúÏö©ÎêòÍ∏∞Εº Í∏∞ÎåÄÌïòΩ∞, ÏÑÝÌñâ Ïó∞͵¨Îêú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÏùò Î∂ÑÏûêÏúÝÏÝÑÌïôÏÝÅ Ïó∞͵¨ Í≤∞Í≥ºÏùò ÎåÄÎ∂ÄÎ∂ÑÏù¥ Í≤ΩÍ∏∞ÎèÑÏôÄ Ï∂©Ï≤≠ÎèÑ ÏßÄÏó≠ÏóêÏÑú ÎåÄÌïú Í≤∞Í≥ºÏûÑÏùÑ Í≥ÝÎݧÌñà ÏùÑ Îïå ÏòÅÎÇ®ÏßÄÏó≠ÏóêÏÑú Ï∂úÌÜÝÎêú ÏǨÎûåκàÏùò Ïó∞͵¨ ÏǨΰÄÎùºÎäî ÏÝêÏóê ÏÑú ÏùòÏùòÍ∞Ä ÏûàÎã§. Ïö∞ζ¨ÎÇòÎùºÏóêÏÑú Ï∂úÌÜÝÎêòÎäî Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκà Ïùò Í∞úÏ≤¥ ÏàòÏóê ÎπÑÌï¥ Î∞úÌëúÎêòÎäî Ïó∞͵¨ Í≤∞Í≥ºÎäî ÏÜåÏàòÏóê Î∂àÍ≥ºÌïòÎØÄ Î°ú ÏúÝÏÝÑÌïôÏÝÅ Ìå®ÌÑ¥ÏùÑ ÎπÑ͵êÌïòÍ∏∞ÏóêÎäî ÏûêΣåÏÝÅ ÌïúÍ≥ÑÏѱÏù¥ ÏûàÎã§. Îî∞ÎùºÏÑú Ï°∞ÏÑÝÏãúÎåÄ ÏǨÎûåκàÏóê ÎåÄÌïú ÏßÄÏÜçÏÝÅÏù∏ Î∂ÑÏÑù Ïó∞͵¨Í∞Ä Ïàò ÌñâÎêòÏñ¥Ïïº ÌïòΩ∞, Í≥ºÎ≥ÄÏù¥Î∂ÄÏúÑ ÎøêÎßå ÏïÑÎãàÎùº coding regionsÏóê ÎåÄÌïú Î∂ÑÏÑùÏùÑ Î≥ëÌñâÌïòÏó¨ ÎçîÏö± ÏÝïÌôïÌïú ÌïòÌîåΰúÍ∑∏ΣπÏùÑ Í≤∞ÏÝïÌïòÎäî Í≤ÉÏù¥ ÌïÑÏöîÌïÝ Í≤ÉÏúºÎ°ú ÏǨΣåÎêúÎã§.
사 사
Î≥∏ Ïó∞͵¨Îäî Ψ∏ÌôîÏû¨Ï≤≠ ͵≠ζΩΨ∏ÌôîÏû¨Ïó∞͵¨ÏÜå Ψ∏ÌôîÏúÝÏÇ∞ Ï°∞ÏǨÏó∞ ͵¨ ÏǨÏóÖÏùò ÏßÄÏõêÏùÑ Î∞õÏïÑ ÏàòÌñâÎêòÏóàÏúºÎ©∞, Ïó∞͵¨Î•º ÏúÑÌï¥ ÏòõÏǨÎûå κà ÏûêΣåΕº ÏÝúÍ≥µÌï¥ Ï§Ä (Ïû¨)Ìïú͵≠Ψ∏ΨºÏó∞͵¨ÏõêÏóê Í∞êÏǨÎìúζ∞Îã§.
REFERENCES
Andrews,
R.M.,
Kubacka,
I.,
Chinnery,
P.F.,
Lightowlers,
R.N.,
Turnbull,
D.M. and
Howell,
N.,
1999,
Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA.
Nature Genetics,
23,
147.


Cho,
E.M.,
Kang,
S.,
Kwon,
E.S. and
Jee,
S.H.,
2010,
Genetic characterization of microorganism from human remains in the Joseon Period.
Conservation Studies,
31,
69-77.
[in Korean with English abstract].
Derenko,
M.,
Malyarchuk,
B.,
Grzybowski,
T.,
Denisova,
G.,
Dambueva,
I.,
Perkova,
M.,
Dorzhu,
C.,
Luzina,
F.,
Lee,
H.K.,
Vanecek,
T.,
Villems,
R. and
Zakharov,
I.,
2007,
Phylogeographic analysis of mitochondrial DNA in northern Asian populations.
The American Journal of Human Genetics,
81,
1025-1041.


Ha,
D.,
2011,
A study on the status of the victims of the Silla sacrificial burials at Imdang.
Journal of the Korean Archaeological Society,
79,
175-204.
[in Korean with English abstract].
Hong,
S.B.,
2014,
Forensic genetic characterization of autosomal 23 STR markers and mtDNA variation in the Korean population. Ph. D. dissertation, Dankook University, Cheonan, 32
Hong,
S.B.,
Kim,
K.C. and
Kim,
W.,
2014,
Mitochondrial DNA haplogroups and homogeneity in the Korean population.
Genes & Genomics,
36
(5),
583-590.

Hong,
S.B.,
Kim,
K.C. and
Kim,
W.,
2015,
Population and forensic genetic analyses of mitochondrial DNA control region variation from six major provinces in the Korean population.
Forensic Science International: Genetics,
17,
99-103.


Jee,
S.H.,
Kim,
Y.J.,
Chung,
Y.J.,
Seo,
M.S. and
Pak,
Y.J.,
2008,
A paleogenetic analysis of human skeletal remains from the Myeongam-ri site, Asan in Korea.
Journal of Conservation Science,
23,
81-93.
[in Korean with English abstract].
Jin,
H.J.,
Tyler-Smith,
C. and
Kim,
W.,
2009,
The peopling of Korea revealed by analyses of mitochondrial DNA and Y-chromosomal markers.
PLoS One,
4,
1
e4210.



Jin,
H.J.,
Kim,
K.C. and
Kim,
W.,
2015,
Phylogenetic analysis of two haploid markers of 500-years-old human remains found in a central region of Korea.
Genes Genomics,
37,
1
33-43.

Kang,
S.,
Kwon,
E.S.,
Moon,
E.J.,
Cho,
E.M.,
Seo,
M.S.,
Kim,
Y.J. and
Jee,
S.H.,
2010,
Usefulness of biochemical analysis for human skeletal remains assigned to the Joseon dynasty in Oknam-ri site in Seocheon, Korea.
Journal of Conservation Science,
26,
1
95-107.
[in Korean with English abstract].
Kim,
Y.J.,
Kim,
S.H.,
Cho,
E.M. and
Lee,
J.W.,
2015,
Mitochondrial DNA analysis of human skeletal remains excavated from Myungam-ri site in Asan, Korea.
Conservation Studies,
36,
33-48.
[in Korean with English abstract].
Kong,
Q.P.,
Yao,
Y.G.,
Liu,
M.,
Shen,
S.P.,
Chen,
C.,
Zhu,
C.L.,
Palanichamy,
M.G. and
Zhang,
Y.P.,
2003,
Mitochondrial DNA sequence polymorphisms of five ethnic populations from northern China.
Hum. Genet,
113,
5
391-405.


Lee,
J.J.,
Ha,
D.R.,
Pak,
S.,
Woo,
E.J.,
Lee,
C.K.,
Kim,
D.H.,
Kim,
J.I. and
Han,
Y.H.,
2008,
The relations of the dead: Identifying the relationship of individuals buried at Imdang, Gyeongsan, through the analysis of mitochondrial DNA from human skeletal remains interred in large mounded tombs.
Journal of the Korean Archaeological Society,
68,
128-155.
a
[in Korean with English abstract].
Lee,
H.Y.,
Song,
I.,
Ha,
E.,
Cho,
S.B.,
Yang,
W.I. and
Shin,
K.J.,
2008,
mtDNAmanager: A web-based tool for the management and quality analysis of mitochondrial DNA control-region sequences.
BMC Bioinformatics,
9,
483.
b



Lee,
H.Y.,
Yoo,
J.E.,
Park,
M.J.,
Chung,
U. and
Shin,
K.J.,
2006,
Mitochondrial DNA control region sequences in Koreans: identification of useful variable sites and phylogenetic analysis for mtDNA data quality control.
International Journal of Legal Medicine,
120
(1),
5-14.


Lee,
K.S.,
Chung,
Y.J.,
Han,
S.H.,
Lee,
M.H.,
Han,
M.S. and
Choi,
D.H.,
1999,
The genetic analysis on ancient human bone in jar coffin excavated from Naju Bokam-ri 3rd tumulus.
Conservation Studies,
20,
5-20.
[in Korean with English abstract].
Maruyama,
S.,
Minaguchi,
K. and
Saitou,
N.,
2003,
Sequence polymorphisms of the mitochondrial DNA control region and phylogenetic analysis of mtDNA lineages in the Japanese population.
International Journal of Legal Medicine,
117
(4),
218-225.


Park,
J.H.,
Cheong,
C.S.,
Jeong,
Y.J. and
Lee,
J.J.,
2015,
Strontium isotope geochemistry of archaeological human tooth enamel excavated from Jeonju, southwestern Korea.
Geosci. J,
19
(2),
185-188.

Rohland,
N. and
Hofreiter,
M.,
2007,
Ancient DNA extraction from bones and teeth.
Nature,
2
(7),
1756-1762.

Seo,
M.S.,
Cho,
E.M.,
Kim,
Y.J.,
Kim,
S.H. and
Kang,
S.,
2014,
The genetic analysis study of ancient human bones excavated at Janggi-dong site, Gimpo.
Journal of Conservation Science,
30
(4),
409-416.
[in Korean with English abstract].

Shin,
J.Y. and
Lee,
J.J.,
2009,
Tracing status-dependent dietary differences in the Silla period through stable isotope palaeodietary reconstruction from Imdang, Gyeongsan.
Journal of the Korean Archaeological Society,
70,
84-109.
[in Korean with English abstract].
Shin,
J.Y.,
Choi,
H.G. and
Lee,
J.J.,
2015,
The socioeconomy of the mid-to-late Joseon dynasty, recorded in stable isotopes of human bone collagen from Honam province sites in Korea.
Journal of the Korean Archaeological Society,
97,
208-231.
[in Korean with English abstract].
Tanaka,
M.,
Cabrera,
V.M.,
Gonz√°lez,
A.M.,
Larruga,
J.M.,
Takeyasu,
T.,
Fuku,
N.,
Guo,
L.J.,
Hirose,
R.,
Fujita,
Y.,
Kurata,
M.,
Shinoda,
K.,
Umetsu,
K.,
Yamada,
Y.,
Oshida,
Y.,
Sato,
Y.,
Hattori,
N.,
Mizuno,
Y.,
Arai,
Y.,
Hirose,
N.,
Ohta,
S.,
Ogawa,
O.,
Tanaka,
Y. and
Kawamori,
R.,
2004,
Mitochondrial genome variation in eastern Asia and the peopling of Japan.
Genome Res,
14,
1832-1850.



The Korea Archaeology and Art History Research Institute
2012,
The Joseon dynasty cemetery in Hwamyeong-dong site,
Busan
13-153.
(in Korean)
Weissensteiner,
H.,
Pacher,
D.,
Kloss-Brandstätter,
A.,
Forer,
L.,
Specht,
G.,
Bandelt,
H.J.,
Kronenberg,
F.,
Salas,
A. and
Schönherr,
S.,
2016,
HaploGrep 2: mitochondrial haplogroup classification in the era of high-throughput sequencing.
Nucleic Acids Res,
58-63.
Willerslev,
E. and
Cooper,
A.,
2005,
Ancient DNA.
Proceeding of the Royal Society,
3-16.
Yonsei DNA Profiling Group
2017,
Monoplex PCR sets for degraded DNA.
http://forensic.yonsei.ac.kr/ (November 6, 2017)